Pflanzenphysiologie
- DNA-Extraktion -
- Versuch 1: Nukleinsäuren 1: Präparation aus pflanzlichem Material und PCR-Amplifikation spezifischer Genregionen
- Versuch 2: Nukleinsäuren 2: Agarose-Gelelektrophorese und Indikatorgene in transgenen Pflanzen
Einleitung:
Nukleinsäuren sind
heteropolymere Verbindungen,
bestehend aus linear aufeinander abfolgenden, monomeren Einheiten,
die als Nukleotide bezeichnet werden.
Nukleotide bestehen aus einer oder mehreren mit Ribose verknüpften
Phosphatgruppen, sowie aus jeweils einem, mittels N-glykosidischer Bindung an die Ribose gebundenen Molekül aus der Gruppe der stickstoffhaltigen,
basisch reagierenden Pyrimidine oder Purine.
Als typische Nukleinsäuren der Lebewesen unterscheidet man DNS (Desoxyribonukleinsäure)
und RNS (Ribonukleinsäure).
Meist werden in der deutschsprachigen Literatur jedoch die im angelsächsischen Sprachgebrauch üblichen Abkürzungen DNA (für engl. deoxyribonucleic acid)
und RNA (für engl. ribonucleic acid) verwendet.
In den Nukleotiden von natürlich auftretenden, polymeren Nukleinsäure-Molekülen treten die Pyrimidin-Basen Cytosin, abgk. mit dem Grossbuchstaben C,
Thymin, abgk. mit T, und Uracil, abgk. mit U,
sowie die Purine Adenin, abgk. mit A,
und Guanin, abgk. mit G, auf.
Dabei kommt Thymin ausschliesslich in der DNA und Uracil (an Stelle des Thymins) ausschliesslich in RNA-Molekülen vor.
Nukleinsäuren können als einzelne, lineare Moleküle ("Einzelstrang") auftreten oder aus zwei, i.d.R. schraubig miteinander verdrillten Einzelsträngen ("Doppelstrang") bestehen.
Die verdrillte Doppelstrang-Form ist insb. typisch für die DNA des Erbguts der meisten Organismen.
Hierbei bilden gegenüberliegende Nukleotide zweier einzelsträngiger DNA-Moleküle untereinander sog. Wasserstoffbrückenbindungen aus und
verbinden so die beiden Einzelstränge zu einem strickleiterförmigen Duplex-Molekül.
Die Ausbildung der Wasserstoffbrücken erfolgt so, dass zwischen den Basen Adenin und Thymin zwei und zwischen den Basen Guanin und Cytosin drei Wasserstoffbrücken entstehen,
was zur Folge hat, dass nahezu ausschliesslich Basenpaarungen in Form von A-T- und G-C-Paaren in einem doppelsträngigen DNA-Molekül auftreten.
Aufgrund der Asymmetrie der Basenpaarungen spricht man bei einem solchen doppelsträngigen DNA-Molekül auch von einem Heteroduplex.
Die Einzelstränge solcher Heteroduplex-Moleküle sind aufgrund der strukturellen Spannungen in den Bindungen der einzelnen molekularen Bestandteile i.d.R. schraubig umeinander gewunden,
so dass ein derartiges DNA-Molekül auch als DNA-Helix (von grch. helix, dt. Windung, Spirale, Kreislauf u.a.) bezeichnet wird.
Die helikale Struktur solcher doppelsträngiger DNA wird in einem Modell beschrieben, das nach den erstbeschreibenden Wissenschaftlern als Watson-Crick-Modell benannt wurde.
In der Serie von aufeinander abfolgenden Nukleotiden in einem Einzelstrang (Sequenz) von DNA und RNA wird eine Gruppe von drei aufeinander abfolgenden Nukleotiden als Basentriplett oder auch als Codon bezeichnet.
Diese Codons bilden den sogenannten genetischen Code,
in dem jedes der 64, aus den Kombinationsmöglichkeiten der vier Basen resultierenden Basentripletts für die Übersetzung in eine der 20 Standard-Aminosäuren codiert oder
eine Regulationsfunktion besitzt.
Der Genetische Code ist weitestgehend universell, d.h. in fast allen Lebewesen codieren die gleichen Codons für die gleichen Aminosäuren (s. hierzu auch die Tabelle Aminosäuren und ihr genetischer Code).
Ausnahmen mit Abweichungen in der Bedeutung der Codons existieren insb. bei den Mitochondrien, aber auch in einigen besonderen Organismen.
Die Codons sind wiederum in grösseren Einheiten zusammengefasst und zu Genen strukturiert,
welche somit für eine Abfolge von Aminosäuren in Form von Peptiden bzw. Proteinen codieren und
deren Aktivität durch verschiedene Regulationsprozesse (Genexpression) gesteuert werden.
Die Peptide und Proteine der Zellen entstehen dabei durch einen komplexen, mehrstufigen Prozess,
bei dem die Codons eines Gens zunächst im Vorgang der Transkription von einem enzymatischen Apparat in eine sog. Boten-RNA (engl. messenger-RNA, abgk. mRNA) übersetzt werden.
Die Sequenz der Codons in den mRNA's kann anschliessend durch den Mechanismus der Translation an den Ribosomen in eine
Abfolge von Aminosäuren umgewandelt werden.
Neben den für Peptide und Proteine codierenden mRNA's werden durch Transkriptionsvorgänge weitere RNA-Moleküle gebildet.
Hierzu zählen u.a. Moleküle der engl. transfer RNA (abgk. tRNA),
die Aminosäure-Moleküle an den Ribosomen übertragen,
die engl. ribosomal RNA (abgk. rRNA) mit strukturell-enzymatischen Funktionen am Ribosom oder
weitere, engl. als cytoplasmic RNA (abgk. cRNA) oder nuclear RNA (abgk. nRNA) bezeichnete RNA-Moleküle mit meist regulativen Funktionen.
Die DNA stellt, abgesehen von RNA-Viren (Retroviren), deren Erbinformation in einzel- oder doppelsträngiger RNA codiert sein kann, die Erbsubstanz des Lebens dar und ist damit Bestandteil aller zellulären Organismen, sowie vieler Viren.
Bei eukaryontischen Organismen ist die DNA
in einem von einer Biomembran begrenzten
Kompartiment,
dem Zellkern oder Nucleus,
lokalisiert, während bei den Prokaryonten die DNA zwar räumlich konzentriert
aber doch mehr oder weniger frei verteilt im Cytoplasma vorliegt.
Die eukaryontische DNA des Zellkerns ist mit Proteinen, den basischen Histonen und
sauren Nicht-Histonen, assoziert.
Durch diese Verbindung mit einem Protein-Gerüst werden besondere Strukturen ("Überstrukturen") realisiert, welche die ursprüngliche Länge der DNA-Moleküle extrem verkürzen,
so dass eine Verpackung von bis zu 2 mm langen DNA-Molekülen im Nucleus, der nur einen Durchmesser von wenigen μm besitzt, möglich wird.
Die mit Proteinen assoziierte DNA wird auch als Chromatin bezeichnet und
ist von 'freier', nicht mit Proteinen assoziierter DNA zu unterscheiden.
Die grösste Verkürzung (Kondensation) der DNA wird während der Vorgänge der Zellteilung (Mitose) erreicht,
wo die Organisation der eukaryontischen DNA in den Überstrukturen der Chromosomen deutlich zutage tritt.
Die Gesamtheit der in Chromosomen organisierten DNA einer Zelle wird als Genom bezeichnet und
ist in allen Zellen eines Organismus, bis auf die Keimzellen oder Zellen, in denen eine spontane Mutation stattgefunden hat, oder die durch transformierende (s.u.) Viren-DNA verändert wurden, identisch.
Prokaryontische DNA ist meist ringförmig strukturiert und häufig finden sich neben der hauptsächlichen Erbinformation weitere codierende Elemente,
die sogenanten Plasmide.
Die Gesamtheit der Erbinformationen der Plasmide wird auch als Plasmon bezeichnet.
Die DNA-Moleküle der Plasmide sind meist ringförmig geschlossen (circuläre DNA) und können in unterschiedlicher Anzahl im Cytoplasma prokaryontischer Zellen auftreten.
Auch die DNA der Plasmide enthält Gene, jedoch sind die von plasmidialen Genen codierten Genprodukte i.d.R. für den Organismus nicht überlebensnotwendig,
können aber entscheidende Funktionen, wie etwa Antibiotika-Resistenzen beisteuern.
In der sogenannten Endosymbiontentheorie geht man davon aus, dass die Herkunft der Zellorganellen der Eukaryonten aus endosymbiotisch in eine Vorläuferzelle aufgenommenen Prokaryonten herrührt.
Dieser Theorie entsprechend verfügen Mitochondrien und Chloroplasten
über eine eigene, meist circuläre DNA, deren Gene im Falle der Mitochondrien für Enzyme
der Atmungskette und im Falle der Chloroplasten für Enzyme des Photosyntheseapparates codieren.
Bei dem gesamten mitochondrialen oder plastidären Erbgut eines Organsimus spricht man auch von einem Chondriom (Mitochondrien) bzw. einem Plastom (Chloroplasten).
Ein Grossteil der ursprünglich vorhandenen Gene dieser Organellen ist jedoch im Laufe der Evolution in die DNA des Nucleus verlagert worden und
man nimmt auch an, dass die Kontrolle der Teilungsaktivität der Organellen, sowie Teile der Genregulation, nucleären Genen unterliegt.
In einer typischen Pflanzenzelle findet sich also im Nucleus, sowie in den Chloroplasten und Mitochondrien DNA, während RNA,
in ihren hauptsächlichen Formen von mRNA, tRNA und rRNA, nahezu überall in der Zelle vorkommen kann.
Die Gesamtmenge an DNA eines Organismus, also dessen Genomgrösse lässt sich zwar als Molekulargewicht angeben,
gebräuchlich ist aber eine Angabe der Anzahl der Basenpaarungen bezogen auf einen Einzelstrang, als die grösstmögliche Menge nicht-renundanter DNA.
Dieser Wert wird auch als C-Wert (engl. c-value) bezeichnet und bietet den Vorteil,
aus der Menge der Basenpaarungen die Anzahl möglicher Gene abzuschätzen.
Einige Werte von Genomgösse und Anzahl der darin vorhandenen Gene ist in Tabelle 1 wiedergegeben.
| Organelle/Organismus | Genomgrösse | Anzahl Gene |
|---|---|---|
| Mitochondrium Tier | 20 kB [2] | 37 (Mensch) [4] |
| Mitochondrium Pflanze | 100-10000 bp [2] | |
| Chloroplast | 120-180 kb [2] | ca. 60-100 [5] |
| Escherichia coli | 4,0 Mbp [2] | ca. 5000 [6] |
| Caenorhabitis elegans | ca. 100 Mbp [7] | ca. 18400 [7] |
| Arabidopsis thaliana | 157 Mbp [8] | ca. 27000 [8] |
| Physcomitrella patens | 480 Mbp [9] | |
| Fritillaria assyrica | 130 Mbp [9] | |
| Homo sapiens | ~ 3,2 Gbp [9] | ca. 20.000-25.000 [10] |
In der Gentechnologie macht man sich den Umstand zunutze, dass Zellen unter bestimmten Vorraussetzungen in der Lage sind, nicht zum Genom gehörige DNA extrazellulärer Herkunft in ihr Genom aufzunehmen (Insertion) und bei entsprechender Struktur der inserierenden DNA die darauf codierte Information auch zu prozessieren, d.h. Genexpression der inserierenden DNA-Sequenz stattfinden zu lassen. Dieser Vorgang wird als Transformation bezeichnet. Natürlicherweise finden solche Transformationsprozesse bei Pflanzen durch Infektion mit dem Bakterium Agrobacterium tumefaciens statt. Dabei überträgt das Bakterium DNA eines sog. Ti-Plasmids in eine verletzte Pflanzenzelle. Von dieser Plasmid-DNA inseriert ein bestimmter Abschnitt, die sogenannte T-DNA, in das pflanzliche, nucleäre Genom, wobei die auf der T-DNA liegenden Gene auch prozessiert, d.h. genetisch exprimiert werden. Durch geeignete gentechnische Verfahren lässt sich dieser Vorgang zur gezielten Veränderung eines pflanzlichen Zielgenoms mit willkürlich gewählten Genen nutzen, die in den Abschnitt der T-DNA des Ti-Plasmids mittels Restriktionsenzymatik inseriert wurden. [1], [2], [3]
Versuchsbeschreibung:
Dieser Versuch gliederte sich in 3 Versuchsteile:
A: CTAB-Präparation von Nukleinsäuren
- Im Versuchsteil A sollten Nukleinsäuren aus pflanzlichem Material bekannter, aber frei gewählter Herkunft extrahiert und isoliert werden.
B: Photometrie von DNA
- In diesem Versuchsteil sollte die DNA-Konzentration in der aus Versuchsteil A gewonnenen Probe photometrisch bestimmt werden.
C: Amplifikation von ausgewählten DNA-Regionen mit Hilfe der PCR
- In diesem Versuchsteil sollte mittels PCR, aus der in Versuchsteil A gewonnenen DNA, die mitochondriale Genregion cox3 und die
chloroplastidäre Genregion psbE amplifiziert werden.
Versuchsdurchführung:
Versuchsteil A:
Ca. 1 g zerkleinertes Pflanzenmaterial von Amorphophallus titanium (Familie: Araneae) wurde in einem Mörser mit vorgelegtem flüssigen
Stickstoff zerstossen und kleingerieben. Von dem erhaltenen Pflanzenpulver wurde 2-3 kleine Spatelspitzen in ein Eppendorf-Cap mit 1 ml vorgewärmter
CTAB-Lösung (Zusammensetzung s.u.) gegeben, mehrfach umgeschüttelt und dann für 5 min. bei 55°C im Wasserbad inkubiert.
Unter dem Abzug wurde 1 ml Chloroform/Isoamylalkohol (24:1 v/v) zugegeben und bei 5000 rpm zentrifugiert. Nach der Phasentrennung wurde die obere, wässrige
Phase mit einer Pipette abgenommen, in ein neues Eppendorf-Cap überführt und die Prozedur der Chloroformierung und anschliessenden Zentrifugation
wiederholt. Dem nun erhaltenen Überstand wurde 1 ml eiskaltes Isopropanol zugesetzt, umgeschwenkt und für 5 min auf Eis inkubiert. Anschliessend
wurde die Probe bei 13000 rpm bei 4°C 10 min zentrifugiert. Der Überstand über dem gebildeten Niederschlag (Pellet) wurde abgenommen und das Pellet
mit 500 μl 70% Ethanol versetzt und wiederum zentrifugiert. Dieser Schritt wurde noch einmal wiederholt, dann wurde das Ethanol abgenommen
und das Pellet in einer Vakuumzentrifuge (SpeedVac) getrocknet und hernach mit 200 μl TE-Puffer (Zusammensetzung s.u.) versetzt.
- 2% (w/v) Cetyltrimethylammoniumbromid, Detergenz zur Denaturierung des Proteinanteils und Auflösung der Membranen
- 1,4 M NaCl, Ionenpufferung insb. durch Natrium, da einwertige Ionen die DNA in Lösung halten
- 20 mM EDTA, Komplexbildner zur Bindung zweiwertiger Ionen
- 100 mM Tris-HCl, pH 8, Puffer zur Einstellung des pH-Wertes
- 2 mM DTT (1,4-Dithiothreitol), Denaturierung von Proteinen durch Auflösung von Schwefelbrücken
- 1 % PVP (Polyvinylpyrrolidon), hochmolekulares Polymer zur "Beschwerung" der Lösung
- 10 mM Tris-HCl, pH 8 (Tris[hydroxymethyl]aminomethan)
- 1 mM EDTA (Ethylendiamintetraessigsäure)
Versuchteil B:
Von der in Versuchsteil A gewonnenen Probe wurden 100 μl in eine Küvette überführt und diese in ein Photometer eingesetzt.
Es wurde die Extinktion bei 260 nm gemessen.
Versuchsteil C:
Für die PCR wurden zwei 25 μl Ansätze nach dem folgenden Schema pipettiert:
- 13,5 μl Aqua bidest.
- 1 μl des Probenmaterials
- 5 μl 5x Puffer für die Taq DNA-Polymerase
- 1 μl 25 mM MgCl2
- 2 μl 2,5 mM dNTP-Mischung
- 1,5 μl der jeweiligen Primer-Mischungen (ein Ansatz mit cox3- und ein Ansatz mit psbE-Primern)
- 1 μl Taq-Polymerase (1 U)
Die PCR-Ansätze wurden in den PCR-Thermo-Cycler eingesetzt, der mit folgenden Parametern programmiert wurde:
- 5 min 95°C Denaturierung genomische DNA
- 30 s 95°C Denaturierung neu hybridisierter/polymerisierter DNA
- 30 s 50°C Hybridisierung der Primer (Annealing)
- 30 s 72°C Synthese 72°C (Elongation)
- 5 min 72°C Synthese aller Stränge bis zum Ende
- Kühlung bei 4°C
Ergebnis:
Versuchsteil A: Es wurde ein milchig-weisses Pellet gewonnen.
Versuchsteil B: Die Ergebnisse der photometrischen Untersuchung ist in der nachfolgenden Tabelle wiedergegeben:
| Species | Quotient 260nm/280nm | Konzentration DNA (ng/μl) |
|---|---|---|
| Cannabis sativa | 1,82 | 94,3 |
| Ginko biloba | 1,80 | 62,2 |
| Amorphophallus titanium | 1,69 | 22,7 |
Versuchsteil C: Die Ergebnisse dieses Versuchsteils werden im Versuchsteil A des Versuches 2 dargestellt.
Ergebnisdiskussion:
Versuchsteil A und B: Wie die Ergebnisse der photometrischen Untersuchung zeigen, konnte aus allen drei Pflanzen ausreichend DNA gewonnen werden, d.h. das in Versuchsteil A gewonnene Pellet bestand tatsächlich zu einem gewissen Anteil aus DNA. Eine Angabe in Relation zur Ausgangsmenge des eingesetzten Pflanzenmaterials ist nicht möglich, da die zur DNA-Extraktion eingesetzte Menge nicht abgewogen wurde. Zu der Probe aus Amorphophallus titanium ist anzumerken, dass die photometrische Untersuchung unverdünnt erfolgte, da eine Messung in Verdünnung negative Werte ergab. Dementsprechend ist, wie aus der Tabelle ersichtlich, die erhaltene DNA-Konzentration von Amorphophallus wesentlich geringer als bei den anderen Proben. Der Quotient aus der Extinktion bei 260 nm und der Extinktion bei 280 nm gibt den Reinheitsgehalt der gewonnenen DNA in Relation zum Proteingehalt der Probe wieder, da Proteine aufgrund der statistisch vorhandenen aromatischen Aminosäuren Tyrosin, Tryptophan und Phenylalanin (evt. Histidin, Aromatizität umstritten) ein Absorptionsmaximum bei 280 nm aufweisen (Peptidbindungen bei 230 nm). Bei den ersten beiden Proben (C. sativa u. G. biloba) liegt der Quotient im Toleranzbereich von 1,8 - 2,0, bei der A. titanium ein wenig darunter.
Aufgaben:
Wieviel μg eines 500 bp langen PCR-Produktes lassen sich theoretisch mit je 30 pmol der Primer amplifizieren, wenn kein anderer
Faktor limitierend ist?
Da unter gegebenen Bedingungen nur die Menge der eingesetzten Primer limitierend wirkt, lässt sich auch nur 30 pmol des PCR-Produktes
herstellen. Veranschlagt man ein durchschnittliches Molekulargewicht von 300 u pro Nucleotid, ergibt sich für ein 500 bp-Abschnitt
eine Molmasse von 300.000 g/mol, welches multipliziert mit 30 pmol eine Masse von 9 μg ergibt, was wiederum der maximal erhältlichen
Menge des PCR-Produkts entspricht.
Versuchsbeschreibung:
Dieser Versuch gliederte sich in zwei Versuchsteile:
A: Gel-Elektrophorese
- Die in Versuchsteil C des Versuchs 1 hergestellten PCR-Produkte, sowie die extrahierten Nukleinsäuren wurden in einem Agaraose-Gel
elektrophoretisch aufgetrennt
B: Indikatorgenaktivität in transgenen Pflanzen
- Für das GUS-Gen transgene Arabidopsis-Keimlinge mit verschiedenen Promotoren, sowie der Wildtyp, wurden zum Nachweis der Genaktivität
der Transgene mit einer colorigenen Lösung (Indikatormedium) versetzt.
Versuchsdurchführung:
Versuchteil A: Je 15 μl der in Versuchsteil C des Versuchs 1 hergestellten PCR-Produkte, sowie 10 μl der extrahierten Gesamt-Nukleinsäure wurden zu 5 μl Ladepuffer (Zusammensetzung s.u.) in ein Eppendorf-Cap gegeben, gut vermischt und in die Geltaschen ('Slots') eines vorbereiteten, mit Ethidiumbromid versetzten, 0,8 %igem Agarosegels eingefüllt. Zusätzlich wurde eine Längenstandard aufgetragen. Dem Gel wurde ein elektrischen Feld von 90 V für ca. 90 min angelegt. Nach Ende der Gellaufzeit wurde das Gel entnommen und unter UV-Strahlung photographiert.
Versuchsteil B: Es wurden drei Ansätze von Keimlingen von Arabidopsis thaliana hergestellt, indem einmal hinsichtlich des GUS-Gens transgene Pflänzchen mit dem CaMVS35 Promotor, einmal GUS-Gen transgene Pflänzchen mit dem Promotor eines Gens für einen Magnesium-Rezeptor und Keimlinge des Wildtyps in Eppendorf-Caps eingefüllt wurden, die eine colorigene Substratlösung enthielten (Zusammensetzung s.u.). Die Ansätze wurden bei 37°C für ca. 1 h inkubiert, die Keimlinge entnommen und photographiert.
Zusammensetzung 3x Lade-Puffer:- 13% Ficoll 400 (w/v), "Beschwerung der DNA"
- 0,04% Kresolrot (w/v), Farbstoff zur optischen Kontrolle des Gellaufs
- pH 7,0 in 100 mM Natriumhydrogenphophat-Puffer
- 10 mmol/l Na2EDTA × 2 H2O
- 3 mmol/l K4[Fe(CN)6] × 3 H2O
- 0,5 mmol/l K3[Fe(CN)6]
- 1 mmol/l X-GlcA (in 10 μl/mg DMF)
- 0,1 % Triton X-100 (v/v)
Ergebnis:
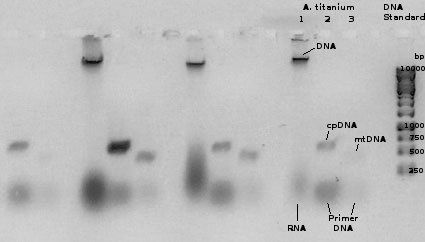
Versuchsteil A: Die Ergebnisse der Gelelektrophorese sind auf dem folgenden Photo abgebildet:
Versuchsteil B: Die Ergebnisse der Nachweisreaktion der Indikatorgene sind auf dem folgenden Photo abgebildet:
Versuchsteil A:
Wie aus der Abbildung 1 ersichtlich, ist die PCR der mitochondrialen cox3-Genregion,
die für eine Untereinheit der Cytochrom-Oxidase codiert, und der chloroplastidären psbE-Genregion,
die für eine Untereinheit des Photosystems II codiert, erfolgreich verlaufen.
In der mit 1 markierten Spur wurde Gesamtnukleinsäure aufgetragen und man sieht im oberen Teil eine deutliche Bande der genomischen DNA,
die deutlich über dem grössten Längenmarker von 10 kbp liegt und damit in etwa im Bereich eines möglichen c-values für A. titanium liegt.
Am unteren Ende des Slot 1 tritt eine diffuse Bande auf ("Wolke", "smear"), deren Ursprung man verschiedenen RNA-Fraktionen zuschreiben muss,
ähnliche diffuse Bandenbildung tritt auch in den Spuren 2 und 3 auf, wobei hier die Herkunft vermutlich überschüssiger Primer-DNA zuzuschreiben ist.
In der Spur 2 war das PCR-Produkt der psbE-Genregion chloroplastidärer DNA aufgetragen und man sieht,
dass hinreichend DNA in einer nicht scharf umgrenzten Bande im Bereich zwischen 500 und 750 bp gebildet wurde.
Ebenso lässt sich, jedoch nur schwach wahrnehmbar,
eine Bandenbildung für das PCR-Produkt der cox3-Genregion mitochondrialer DNA knapp unterhalb des 500 bp Längenstandards erkennen.
Damit liegen die erhaltenen Banden innerhalb der erwarteten Längenbereiche von 580 bp für die psbE-Genregion und 450 bp für die cox3-Genregion.
Abweichende Längen der erhaltenen PCR-Produkte von der erwarteten DNA-Länge können immer dann auftreten, wenn in dem
zu untersuchenden Organismus z.B. eine Insertion in den zu untersuchenden Abschnitt gegeben hat, etwa durch Transposons. Auch abweichende
Primerbindungen, z.B. durch einen weiter entfernt bindenden Primer können für eine abweichende Länge verantwortlich sein.
Erhält man kein PCR-Produkt, ist man gezwungen die PCR mit veränderten Bedingungen (PCR-Zyklen) nochmals durchzuführen oder
u.U. die Extraktion zu wiederholen, da man nicht genug Ausgangsmaterial in der Probe hatte.
Verantwortlich für eine fehlgeschlagene PCR können aber auch fehlerhafte Primer, Nucleotide, Taq-Polymerase oder gar Pipettierfehler sein,
so dass in jedem Fall das Prokoll und die Durchführung der PCR noch einmal überprüft werden sollte.
Eine zu grosse Menge gewonnenen PCR-Materials ist i.d.R. schadlos.
Hierin liegen auch die Vor- und Nachteile der PCR, einerseits kann aus geringen Ausgangskonzentrationen nahezu unbegrenzt DNA gewonnen werden,
aber Vorraussetzung ist zum einen die Kenntnis begrenzender Sequenzen der zu amplifizierenden DNA und andererseits der richtige Einsatz der Reagentien eines PCR-Ansatzes,
insb. sollten die Primer nicht überlappen, nicht zu dicht beieinander liegen oder zu lang sein.
Versuchsteil B: Wie zu erwarten war, zeigt der Wildtyp, ganz links in Abb. 2 keinerlei Blaufärbung, da ja das zur Substratumsetzung notwendige GUS-Gen in diesen Pflanzen nicht vorhanden ist. Das GUS-Gen codiert für das Enzym β-Glucuronidase, welches wiederum in der Lage ist, bestimmte glykosidische Bindungen zu spalten. Dies macht man sich zunutze, indem den Zellen, die dieses Gen exprimieren, ein spezielles colorigenes Substrat (X-Gluc) angeboten wird (z.B. durch Inkubation). Das Glykosid X-Gluc ist das Cyclohexylammoniumsalz der 5-Brom-4-chlor-3-indolyl-β-D-glucuronsäure, d.h. es besteht aus einer Glucuronsäure (einer Zuckersäure) und einem aromatischen Anteil. Bei der Umsetzung mit β-Glucuronidase wird der aromatische Anteil abgespalten und bildet unter Dimerisierung und Sauerstoffeinwirkung einen blauen Farbstoff, der, wenn er in Geweben auftritt, diese charakteristisch anfärbt. Da das natürliche Vorkommen des GUS-Gens (und damit der β-Glucuronidase) hauptsächlich auf die Vertebrata (Wirbeltiere) beschränkt ist, eignet es sich besonders gut zur Anwendung als Reportergen in transgenen Pflanzen, wobei die Stärke und der Ort der Farbstoffbildung in direkter Relation zum verwendeten Promotor des Transgen-Konstruktes steht. So zeigt die transgene Variante mit dem CaMVS35-Promoter in der Mitte der Abb. 2 eine durchgängige Blaufärbung in nahezu allen Gewebeteilen, da der CaMVS35 Promotor konstitutiv, d.h. unabhängig von gewebespezifischer Genregulation, exprimiert wird. Die Probe mit dem Promotor des Magnesiumrezeptors ganz rechts in Abb. 2 zeigt nur im Bereich der Blätter, und hier besonders im Bereich der Blattnerven, eine Blaufärbung, was darauf hindeutet, dass der Magnesiumrezeptor in diesem Stadium der Entwicklung in diesen Geweben exprimiert wird. Somit konnte die Wirksamkeit des transgenen Indikatorgens im Hinblick auf die differentielle, gewebeabhängige Genexpression demonstriert werden. [11]
Aufgaben:Was bedeuten die Begriffe Selbstung, Polyploidie und somaklonale Variation ?
Polyploidie - Genetischer Zustand, der sich dadurch auszeichnet, dass Zellen oder ganze Organismen mit mehr als dem diploiden Chromosomensatz ausgestattet sind.
somaklonale Variation - Abweichungen vom genetischen Ausgangszustand bei Pflanzen, die in-vitro Kulturen angezüchtet werden. Die Ursachen können genetischer oder epigenetischer Natur sein, häufig sind diese auch noch völlig ungeklärt. [12]
Nennen Sie einige Nutzanwendungen transgener Pflanzen, die von Interesse für eine Nutzanwendung sind.
Bt-Mais, eine transgene Maissorte, die mit einem Gen aus Bacillus thuringiensis transformiert wurde, das für ein Toxin codiert (Bt-Toxin), welches giftig für Insekten ist (Darmperforation), so dass Schadinsekten, insb. der innerhalb der Maispflanze sich entwickelnde Maiszüngler, ausgemerzt werden.
Amflora, transgene Kartoffel der Firma BASF Deutschland, die mit Genen transformiert wurde, die die Kartoffel zur Produktion spezieller Polysaccharide befähigt, welche wiederum zur Herstellung von chem. Verbindungen im industriellen Massstab genutzt werden können.
Welche Argumente gegen eine Nutzanwendung transgener Pflanzen erscheinen Ihnen zutreffend ?
Andererseits wird die paradigmatische Ansicht vieler Wissenschaftler und Befürworter der "Grünen Gentechnologie", dass die Aufnahme gentechnisch veränderter Organismen durch die Nahrung keinerlei Auswirkung auf den Zustand des aufnehmenden Organismus hat, bspw. durch eine Veröffentlichung von Hehemann et al. (2010) [13] widerlegt. Diese Untersuchung widerspricht der gängigen Meinung, dass grundsätzlich durch Aufnahme von Nahrung, also auch transgener Nahrung, jedwede Beziehung zum Ausgangsmaterial, insb. der genetischen Information, im Prozess der Verdauung zerstört wird, und legt dar, wie Gene bestimmter Bakterien (Zobellia galactanivorans), die insb. mit Rotalgen (Rhodophyta) assoziiert sind, auf Bakterien des Verdauungstraktes des Menschen im Zuge der Nahrungsaufnahme übertragen werden.
Derartige Vorgänge sind weitestgehend unerforscht und weisen u.U. neue Wege auf, die nicht nur neue Erkenntnisse über die Evolution der mannigfaltigen Darmflora des Menschen liefern können, sondern auch Hinweise auf mögliche und bisher unberücksichtigte Mechanismen des horizontalen Gentransfers geben können. Erhärten sich die Beweise für solche nahrungsgekoppelten Vorgänge des horizontalen Gentransfers, lassen sich auch Übertragungen von gentechnisch in die Nahrung eingebrachten "Fremdgenen", wie z.B. dem Bt-Gen, auf die Darmflora nicht ausschliessen und damit würden gentechnisch veränderte Pflanzen in einem ganz neuen Licht erscheinen, nämlich als potentielle Vektoren genetischer Information.
Ferner sind Einwände unter dem Gesichtpunkt zunehmender Allergien überprüfenswert, da transgene Pflanzen durch die Transformation durchaus neue Eigenschaften aufweisen können, die wiederum zu neuartigen Allergenen führen können. Auch diesem Gesichtspunkt wird nach meinem persönlichen Wissensstand in der wissenschaftlichen Forschung zu wenig Beachtung geschenkt.
Einen weiteren Aspekt, unter den Gesichtpunkten von Ökologie und Evolution, stellt die Tatsache dar, dass man in transgenen Pflanzen Gene aus verschiedenen Species in einem Genpool zusammenbringt, welcher so von der Natur nicht vorgesehen ist. D.h. man könnte mit grossflächiger Ausbringung und Anwendung von gentechnisch veränderten Pflanzen das von der Natur über Milliarden Jahre entwickelte, auch "informationstechnisch" miteinander verwobene Netzwerk der Evolution und der daraus entstandenen Standort-Ökologie empfindlich und vor allen Dingen irreversibel stören. Ein natürliches Beispiel eines "mixing vessels", d.h. die sich vermischenden Gene eines Organismus in einem Wirtsorganismus, stellt bspw. das H1N1-Virus dar, das im Jahre 2009 grassierte. Da Pflanzen, denkt man an das Cadang-Cadang-Virus oder Tomaten-Viroide, anscheindend noch sehr viel empfindlicher und suszeptibler auf frei flukturierende genetische Information reagieren, sind die Wechselwirkungen von transgenen Pflanzen mit solchen natürlichen Vektoren noch weitgehend unerforscht. Zudem können weitere Wechselwirkungen durch koevolutionäre Prozesse entstehen, indem transgene Pflanzen die Resistenz von Viren oder Insekten gegenüber den Genwirkungen des transformierten Erbgutes erhöhen. Ungeklärt sind in diesem Zusammenhang auch noch, v.a. auf internationaler Ebene, die patentrechtlichen, haftungsrechtlichen und versicherungtechnischen Fragen, insb. was potentiell negative Folgen von transgenen Pflanzen angeht. Würde z.B. die Firma Monsanto, wäre sie für die Herausbildung eines "Supervirus" durch transgene Pflanzen ihrer Produktreihe verantworlich, welcher auch andere Pflanzen und die damit verbundenen Ernten schädigen würde, in Haftung genommen ?
Stellt man sich eine globale "Genkrise", analog einer Banken- oder Finanzkrise, durch schlampiges Management, vergessliche Forscher, Computerfehler, Produktionsstörungen etc. vor, wären die Folgen katastrophal, da die Nahrungskette und nicht zuletzt die Zusammensetzung der Atmosphäre vom Gedeihen der Pflanzenwelt abhängen. Insofern kann man getrost behaupten, das in der, im Freiland angewendeten, Gentechnik ein grösseres Riskiopotential als in der Atomtechnik schlummert. Demgegenüber stehen natürlich Argumente einer wachsenden, nach Nahrung verlangenden Weltbevölkerung, Fortschrittsdenken und nicht zuletzt Zwänge der wirtschaftlichen Profitabilität. Insofern gilt es sicherlich sorgfältig zwischen Chancen und Risiken der Gentechnik im allgemeinen, aber im besonderen in der grünen Freiland-Technik, abzuwägen. Ein vielversprechender Ansatz könnte die verpflichtende Anwendung von "Safety"-Techniken sein, die die Vermehrung und Vermischung transgener Pflanzen in natürlicher Umwelt ausschliesst, so dass diese als "genetisch inert" gegenüber dem natürlichen "Hintergrund" erscheinen.
Nehmen Sie an, Sie haben aus einer Protoplastentransformation mit einem Gen, das zu einer roten Blütenpflanze führt, intakte, fertile Gänseblümchen erhalten. Wie wird die F1 aussehen ?
Wie viele Gene essen Sie jeden Tag ?
Referenzen:
[1] Taiz, Zeiger; 'Plant Physiology', 4th Edition, Spektrum Akademischer Verlag 2007
[2] Berg, Singer; 'Gene und Genome', 1st Edition, Spektrum Akademischer Verlag 1992
[3] Richter; Pflanzenphysiologie, 5. Auflage, Thieme Verlag 1988
[4] Wikipedia EN, Mitochondrium
[5] Wikipedia EN, Chloroplast
[6] Wikipedia DE, Escherichia coli
[7] Wikipedia EN, Caenorhabitis elegans
[8] Wikipedia EN, Arabidopsis thaliana
[9] Wikipedia EN, Genome
[10] Wikipedia EN, Homo sapiens
[11] Wikipedia DE, X-Gluc
[12] Wikipedia DE, Somaklonale Variabilität
[13] Hehemann, J.-H., Correc, G., Barbeyron, T., Helbert, W., Czjzek, M., Michel, G. (2010) 'Transfer of carbohydrate-active enzymes from marine bacteria to Japanese gut microbiota.', Nature, 464, 908-914, DOI: 10.1038/nature08937